Rất nhiều quá trình sinh học phân tử được xúc tác bởi hai hay nhiều protein tương tác với nhau như quá trình điều khiển hoạt động của các gen điều khiển, quá trình biệt hóa từ một tế bào thường thành tế bào bệnh….. Việc nghiên cứu mối tương tác giữa các protein có vai trò quan trọng giúp chúng ta hiểu hơn về cơ chế điều khiển hay ức chế hoạt động của một số gene điều khiển cũng như tìm ra nguyên nhân của một số bệnh nan y như bệnh mất trí nhớ (Alzheimer’s disease) hay rối loạn tâm thần Creutzfeldt–Jakob disease. Kiến thức đó có thể được xem như cơ sở để nghiên cứu tạo ra các loại thuốc đặc trị bênh nan y nói trên cũng như trong tạo ra các loại thuốc trừ nấm bệnh mà ít hoặc không có các tác động không mong muốn.
Để dự đoán khả năng tương tác giữa các protein người ta xây dựng một số chương trình tin sinh học như string, genemania … để tính toán khả năng tương tác các protein. Cơ sở dữ liệu của chương trình này cho phép dự đoán các protien có khả năng tương tác với protein quan tâm. Tuy nhiên, tính toán đó chỉ mang tính chất lý thuyết và điều kiện tiêu chuẩn và được phân tích kỹ trên hệ dữ liệu của một số sinh vật mô hình như chuột, ruồi giấm, giun đốt, nấm men …. Trong điều kiện thực tế, tùy điệu kiện môi trường và điều kiện sinh lý khác nhau, tương tác giữa các protein cũng bị thay đổi, hay ở các sinh vật khác nhau, quá trình tương tác cũng khác nhau. Tương tác giữa các protein này có thể bền vững hay tạm thời; mạnh hay yếu tùy thuộc vào từng loại protein và từng điều kiện cụ thể. Tùy theo tính chất của liên kết protein người ta xây dựng những phương pháp phân tích phù hợp như Co-Immunoprecipitation (co-IP) và Pull-Down assay được sử dụng trong phân tích protein tương tác lâu dài và bền vững; Crosslinking Protein Interaction Analysis và Label Transfer Protein Interaction Analysis được sử dụng trong phân tích protein tương tác tạm thời và yếu; và Far-Western Blot Analysis dùng trong phân tích tương tác lâu dài. Trong các phương pháp trên, Pull-Down protein là một phương pháp phân tích protein đơn giản, nó có thể thực hiện ở những phòng thí nghiệm có điều kiện trang thiết bị tối thiểu.
Pull-down protein là một phương pháp nhân tạo (in vitro) được sử dụng để xác định tương tác vật lý giữa một protein đã biết với một hay nhiều protein khác. Phương pháp này có thể sử dụng với 2 mục đích: (1) kiểm tra lại kết quả của một số chương trình tin sinh học dùng dự đoán tương tác protein; (2) sàng lọc các protein có khả năng tương tác với một protein xác định. Phương pháp này về cơ bản giống như phương pháp phân tích tương tác protein khác (Immunoprecipitation) ngoại trừ sử dụng mồi protein thay cho việc sử dụng kháng thể (antibody). Do vậy phương pháp khá đơn giản nhưng lại đặc hiệu cao và rút ngắn thời gian của việc sàng lọc. Trong phương pháp này, một mồi nhử protein (bait protein) được gắn với một gốc kết hợp cố định đặc hiệu cho đuôi như glutathione S-transferase (GST), polyHis hay Biotin…. Cấu trúc này hoạt động như một mồi protein để câu các protein có khả năng bám vào khi được trộn chung với hỗn hợp protein tổng số được tách từ tế bào. Gần đây phương pháp này được tối ưu hóa tạo ra những bộ kít chuyên dùng trong phân tích tương tác protein và được gọi là các bẫy protein như GFP-trap kít của Chromotek. Bẫy này có thể lôi được bất kỳ protein nào và protein tương tác với nó ra khỏi hỗn hợp protein khi protein đó được gắn với GFP, YFP và CFP protein.
Dưới đây là một quy trình ngắn gọn dùng trong sàng lọc protein tương tác với một protein đã biết (ví dụ Tup1 protein của Saccharomyces cerevisiae (S. cerevisiae).
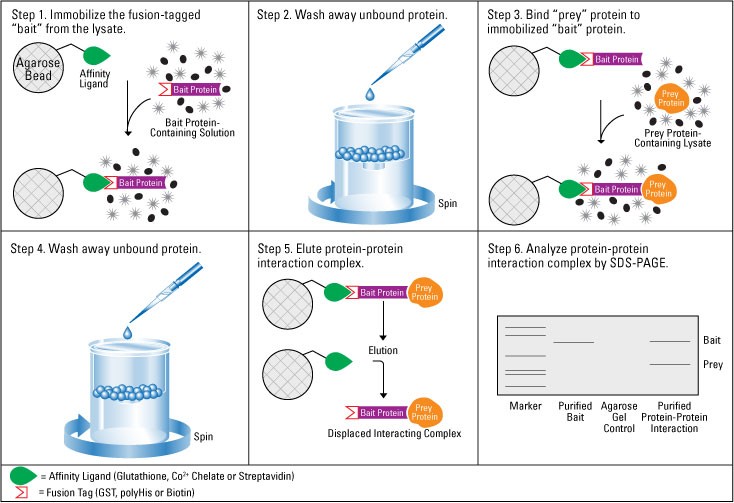
1. Gene mã hóa cho protein quan tâm (Tup1) được gắn với gene mã hóa GFP thông qua một linker giúp liên kết giữa hai protein bền vững và linh động.
2. Cấu trúc trên được chuyển vào nấm men S. cerevisiae, sàng lọc và kiểm tra thể chuyển gene.
3. Nuôi thể chuyển gene nói trên thu sinh khối và tách chiết protein tổng số.
4. Bi GFP-trap của Chromotek được trộn với protein tổng số và ủ trong 1 giờ.
5. Ly tâm loại bỏ dịch nổi và rửa bi GFP-trap với dung dịch đệm thích hợp.
6. Tách protein ra khỏi bi GFP bằng nhiệt độ (95oC) hoặc hòa tan trong pH thấp.
7. Điện di protein thu được trên gel polyacrylamid.
8. Protein trong polyacrylamid gel được cắt với enzyme đặc hiệu như trypsin.
9. Hỗn hợp các peptit thu được sau khi cắt với enzyme đặc hiệu tiếp tục được phân tích bằng phương pháp khối phổ.
10. Kết quả phân tích khổi phổ được xử lý với chương trình phân tích MaxQuant và Perseu, từ đó có thể chỉ ra danh sách những protein có khả năng tương tác với protein quan tâm Tup1.
Tài liệu tham khảo
https://www.thermofisher.com/us/en/home/life-science/protein-biology/protein-biology-learning-center/protein-biology-resource-library/pierce-protein-methods/pull-down-assays.html
http://www.chromotek.com/products/nano-traps/gfp-trap/gfp-trapr-a/
https://www.thermofisher.com/us/en/home/life-science/protein-biology/protein-biology-learning-center/protein-biology-resource-library/pierce-protein-methods/co-immunoprecipitation-co-ip.html
http://www.biochem.mpg.de/226863/Tutorials
Cox, J. and Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat Biotechnol, 2008, 26, pp 1367-72.
Phizicky, E.M., and Fields, S. 1995. Protein-protein interactions: methods for detection and analysis. Microbiological Reviews 59:94-123.
Shevchenko, A., Wilm, M., Vorm, O., and Mann, M. (1996) Mass spectrometric sequencing of proteins from silver stained polyacrylamide gels. Anal. Chem.68, 850-858.
Tin bài: Bùi Tri Thưc - Khoa CNSH&CNTP
Đăng tin: Bùi Đình Lãm

| Đang online | 637 |
| Hôm nay | 2266 |
| Hôm qua | 3860 |
| Tuần này | 29543 |
| Tuần trước | 38389 |
| Tháng này | 3446050 |
| Tháng trước | 5230891 |
| Tất cả | 51666011 |
Lượt truy cập: 51666030
Đang online: 656
Ngày hôm qua: 3860
Phụ trách kỹ thuật: 0987. 008. 333