1. Giới thiệu
CRISPR/Cas9 đang thu hút sự quan tâm của hầu hết các nhà khoa học trên thế giới trong lĩnh vực chỉnh sửa bộ gen và các lĩnh vực sinh học khác. CRISPR/Cas9 được khám phá từ một cơ chế tự vệ trong vi khuẩn, hệ thống này có thể dễ dàng lập trình để cắt và chỉnh sửa bất kỳ trình tự DNA nào ở các loài khác nhau. Do vậy, việc tìm hiểu cơ chế của CRISPR/Cas sẽ là một công cụ công nghệ sinh học nhanh chóng được sử dụng rộng rãi trong việc biến đổi DNA bộ gen.
2. Cấu trúc của CRISPR trong genome vi khuẩn
Nghiên cứu cho thấy tổ chức của CRISPR (Clustered Regularly Interspaced Short Palindromic Repeat -cụm đoạn lặp xuôi ngược ngắn phân cách đều) trong genome vi khuẩn rất đa dạng. Một locus (vị trí) CRISPR cấu thành từ các đoạn lặp (repeats) dài 21 đến 48 bp xen kẽ bởi các trình tự dài 26 đến 72 bp gọi là các đoạn đệm (spacers). Trong một locus thì kích thước của đoạn lặp và đoạn đệm rất ổn định. Mỗi vùng CRISPR được đặc trưng bởi trình tự của đoạn lặp điển hình trong locus đó. Một locus CRISPR chỉ chứa các đoạn lặp và các đoạn đệm, không có khung đọc mở (open reading frame).
.jpg)
Hình 1. Cấu trúc của một vùng CRISPR
Trong một vùng CRISPR có những trình tự đoạn lặp rất bảo thủ. Hầu hết các đoạn lặp có tính palindromic một phần (nghĩa là một phần trình tự xuôi và ngược giống nhau), dẫn đến khả năng hình thành nên cấu trúc thứ cấp ổn định, bảo thủ cao. CRISPR thường nằm trong nhiễm sắc thể, nhưng cũng có trường hợp nằm trong plasmid.
Phía thượng nguồn (up-stream) của một vùng CRISPR là leader region (là trình tự đánh dấu sự bắt đầu của một CRISPR array; CRISPR array là trình tự bao gồm các đoạn đệm và lặp) dài từ 20 đến 534 bp. Vùng CRISPR được phiên mã do một promoter nằm trong leader region. Ngoài ra, luôn tìm thấy vài gene đi kèm CRISPR (CRISPR-associated – Cas, yếu tố đi kèm với CRISPR)ở vùng lân cận, tạo thành hệ thống CRISPR-Cas (bao gồm locus CRISPR và các gene Cas lân cận). Một hệ thống CRISPR-Cas thường gồm 4 đến 20 gene cas khác nhau. Các gene cas mã hóa một họ protein lớn và đa dạng có các domain điển hình của nuclease, helicase, polymerase và các protein gắn polynucleotide. Các gene cas này có thể nằm ở phía thượng nguồn (up-stream) hay hạ nguồn (dow-stream) của vùng lặp/đệm.
.jpg)
|
Hình 2. Một vùng CRISPR tổng quát. Các mũi tên trắng lớn biểu diễn các gene cas phía thượng nguồn của locus CRISPR. L chỉ ra vùng dẫn (leader region), kèm với kích thước có thể của nó. Hình thoi đen (trình tự màu đen trong khung) là các trình tự lặp trong CRISPR1 của Streptococcus thermophilus strain DGCC7710. Các chữ nhật nhỏ tô màu (các trình tự tô màu trong khung) chỉ ra các trình tự khác nhau của đoạn đệm (spacer) có kích thước tương tự có trong một CRISPR.
|
Có thể có nhiều vùng CRISPR trong hệ gene của sinh vật prokaryote. Số lượng lớn nhất phát hiện được cho tới nay là 20 locus CRISPR trong hệ gene của Methanococcus jannaschii. Các đoạn lặp và đệm trong CRISPR có thể chiếm tới 1% tổng trình tự toàn hệ gene của vi khuẩn. Có nhiều suy đoán về vai trò của CRISPR như vai trò trong tái sắp xếp nhiễm sắc thể, điều hòa biểu hiện của các gene lân cận, sửa chữa DNA…
3. Cơ chế hoạt động CRISPR-Cas9 và ứng dụng trong chỉnh sửa hệ gene (genome editing)
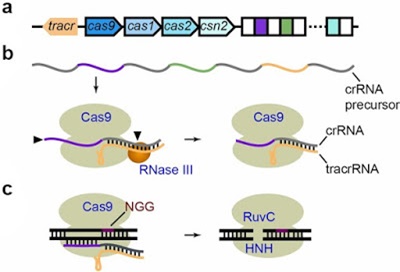
Hình 3. Cơ chế hoạt động của CRISPR-Cas9, hệ thống thuộc kiểu II-A ở Streptococcus SF370 (Bikard and Marraffini, 2013).
(a) Tổ chức của hệ thống CRISPR-Cas9 trong Streptococcus pyogenes SF370. Có 4 gene cas trong hệ thống là cas9, cas1, cas2 và csn2; 3 genes sau được cho là liên quan tới sự tích hợp các trình tự spacer mới vào CRISPR array. Sau các gene cas là một CRISPR array chứa bảy đoạn lặp (màu trắng, dài 36 nt) và sáu đoạn đệm (tô màu, dài 30 nucleotide) có trình tự tương ứng với trình tự của các thực khuẩn thể của loài S. pyogenes. Phía trước các gene Cas là gene mã hóa cho một đoạn RNA nhỏ gọi là trans-activating CRISPR RNA (tracrRNA) có trình tự tương đồng với trình tự của các đoạn lặp.
(b). Quá trình xử lý crRNA. Vùng CRISPR được phiên mã thành một đoạn dài duy nhất (gọi là tiền crRNA) chứa các đoạn lặp (màu xám) và đoạn đệm (các màu khác nhau). Được Cas9 hỗ trợ, tracrRNA tương tác với mỗi trình tự lặp để tạo nên các RNA sợi kép (double-stranded RNA – dsRNA) mà sẽ bị RNA III cắt, tạo ra các crRNA ngắn từ đoạn tiền crRNA dài. Các đoạn crRNA này được xử lý thêm ở đầu 5’, khiến cho trình tự định hướng chỉ còn dài khoảng 20 nucleotide. Mũi tên đen chỉ ra vị trí xảy ra sự xử lý RNA.
(c) Can thiệp đối tượng. Cas9 quét toàn bộ DNA sợi kép của đối tượng xâm nhập để tìm vùng bắt cặp bổ sung với crRNA và tạo ra đoạn cắt trên DNA sợi kép đó bằng cách sử dụng hai domain nuclease riêng rẽ là RuvC-like nuclease ở đầu N (cắt sợi DNA bổ sung), và HNH-like nuclease ở giữa protein (cắt sợi DNA không bổ sung). Yêu cầu phải có một trình tự NGG (gọi là trình tự PAM) ngay phía sau trình tự mục tiêu thì sự cắt mới xảy ra.
Các hệ thống khác cần các protein Cas khác nhau cho bước xử lý crRNA và bước cắt DNA. CRISPR-Cas9 khác biệt ở chỗ nó chỉ cần một protein Cas duy nhất là Cas9 (Jinek, M., et al., 2012).
Dựa trên cấu trúc tự nhiên của CRISPR-Cas9, các nhà nghiên cứu đã đơn giản hóa hệ thống này bằng cách kết hợp tracrRNA và crRNA thành một RNA định hướng duy nhất (single guide RNA – sgRNA). Cas9 định hướng bởi sgRNA được chứng minh là hiệu quả tương đương với Cas9 định hướng bởi tracrRNA và crRNA riêng rẽ. Để phục vụ cho mục đích chỉnh sửa gene, các thành phần không liên quan như vùng dẫn (leader region), các gene Cas có vai trò khác, và các đoạn lặp bị loại bỏ. Gene mã hóa cho Cas9, RNA định hướng (sgRNA) chứa trình tự mục tiêu được kết hợp vào một plasmid biểu hiện nhỏ. Khi chuyển các plasmid này vào tế bào mục tiêu, Cas9 cùng sgRNA được biểu hiện đồng thời. Chúng kết hợp với nhau tạo thành một hệ thống CRISPR-Cas9 đơn giản mà hiệu quả, có thể tạo ra điểm đứt gãy trên DNA tế bào chủ tại vị trí mong muốn.
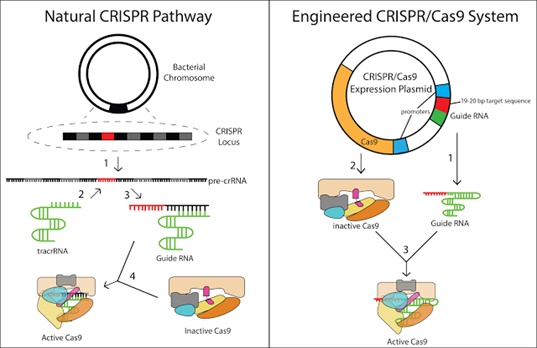
Hình 4. Các hệ thống CRISPR tự nhiên và nhân tạo. Các bước xảy ra với hệ thống CRISPR tự nhiên: 1. Phiên mã tạo ra tiền crRNA và tracrRNA 2. tracrRNA liên kết với crRNA 3. Tạo ra các RNA định hướng từ tiền crRNA 4. nuclease Cas9 đang ở trạng thái bất hoạt gắn kết với RNA định hướng để trở thành Cas9 hoạt hóa. CRISPR nhân tạo: 1. Phiên mã nên RNA định hướng thành một sợi duy nhất 2. Phiên mã và dịch mã tạo nên nuclease Cas9 3. RNA định hướng gắn với Cas9 và hoạt hóa Cas9. (Nguồn: https://sites.tufts.edu/crispr/genome-editing/)
crRNAs dẫn Cas9 có trình tự protein đặc hiệu, nó được ghi nhận thông qua phần bổ sung của crRNA. Enzyme Cas9 nuclease sau đó được đưa vào phá huỷ sợi đôi của DNA mục tiêu. Điểm đứt gãy sợi kép trên DNA có thể được sửa chữa theo hai hướng: non-homologous end-joining (NHEJ – nối đầu cuối không tương đồng) hoặc homology-directed repair (HDR – sửa chữa dựa theo sự tương đồng) (Overballe-Petersen et al., 2013; Gong et al., 2005).
NHEJ thường dẫn tới các đột biến chèn hoặc xóa đoạn nhỏ, khiến cho gene mục tiêu bị bất hoạt. Có thể xóa một đoạn gene tương đối lớn nếu dùng hai crRNA, và có thể tạo ra sự chuyển vị hoặc đảo ngược nhiễm sắc thể nếu dùng crRNA định hướng tới hai nhiễm sắc thể khác nhau (Harrison et al., 2014).
Nếu có một đoạn DNA mẫu (template DNA, hay donor DNA) tương đồng với vị trí mục tiêu thì DSB này có thể được sửa chữa bằng cơ chế HDR. Phần DNA ở quanh vị trí đứt gãy sẽ được thay thế bằng phần DNA tương ứng ở DNA mẫu. Nếu một đột biến được chủ ý đưa vào DNA mẫu thì đoạn DNA sau khi sửa chữa sẽ mang đột biến đó (Hình 5A) (Overballe-Petersen et al., 2013; Gong et al., 2005)
Nhưng ngay cả khi có DNA mẫu thì nhiều khả năng là không chỉ cơ chế HDR, mà cả NHEJ, cùng xảy ra, dẫn tới hiện tượng chèn/xóa đoạn không mong muốn. Một biến thể của Cas9 là Cas9D10A giúp hạn chế điều này. Cas9D10A chỉ có khả năng cắt DNA ở một sợi (chứ không phải ở cả hai sợi như Cas9 thông thường), do đó không kích hoạt cơ chế NHEJ. Khi có mặt DNA mẫu thì việc sửa chữa DNA chỉ được tiến hành theo cơ chế HDR (Cong et al, 2013). Việc sử dụng nhiều hơn một phức hợp Cas9D10A để tạo nên các vị trí đứt gãy sợi đơn (nick) cạnh nhau giúp tăng đáng kể sự đặc hiệu mục tiêu (Ran et al., 2013; Hình 5B).
Một biến thể khác nữa của Cas9 là nuclease-deficient Cas9 (dCas9) (Qi et al., 2013; Hình 5C). không có khả năng cắt DNA). Các đột biến xảy ra với HNH và RuvC domain bất hoạt khả năng cắt DNA, nhưng vẫn giữ lại khả năng liên kết với DNA (Jinek et al., 2012; Gasiunas et al., 2012). Một ứng dụng của biến thể này là biến dCas9 thành công cụ hoạt hóa hoặc ức chế phiên mã khi dung hợp dCas9 với các domain tác động (hoạt hóa hoặc ức chế). Ngoài ra, có thể lợi dụng khả năng bắt cặp đặc hiệu trình tự của dCas9 với DNA để hiển thị trình tự DNA mong muốn trên genome bằng cách dung hợp dCas9 với protein huỳnh quang (Chen et al., 2013).
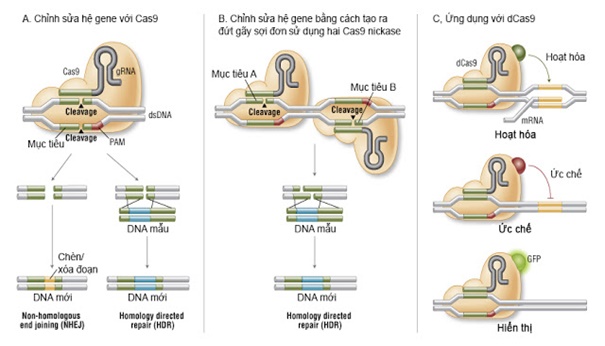
Hình 5. Chỉnh sửa hệ gene bằng hệ thống CRISPR-Cas9 (Gong et al., 2005; Overballe-Petersen et al., 2013; Cong et al., 2013; Qi et al., 2013).
A. Cas9 nuclease dạng tự nhiên có vị trí đặc hiệu giúp cho hoạt động tách DNA sợi đôi thành hai sợi để sửa chữa đứt gãy. Trong trường hợp không có mẫu sửa chữa tương đồng, kết nối không đồng nhất có thể dẫn đến các indels làm gián đoạn chuỗi mục tiêu. Ngoài ra, đột biến chính xác và knock-in có thể được thực hiện bằng cách cung cấp một mẫu sửa chữa đồng thời và khai thác con đường sửa chữa đồng nhất.
B. Đột biến Cas9 xác định ở một điểm đặc hiệu cho nick-sợi đơn. Hai sgRNA có thể được đưa vào để sử dụng phá vỡ sợi đôi mà sau đó có thể hướng đến duy trì sửa chữa đồng nhất.
C. Cas9 thiếu hụt nuclease có thể được kết hợp với các vùng khác nhau của yếu tố ảnh huởng ở vị trí đặc hiệu.
Kết luận
CRISPR/Cas đang ngày càng chứng tỏ tiềm năng ứng dụng mạnh mẽ của mình. Các nhà khoa học đang tiến hành việc nghiên cứu để sử dụng hệ thống này để sửa chữa các sai hỏng trong DNA bộ gen trong các bệnh lý di truyền. Một hướng ứng dụng khác là trong các nghiên cứu tế bào gốc nhằm biệt hóa các tế bào gốc thành nhiều loại tế bào khác nhau nhờ điều chỉnh di truyền trong DNA bộ gen. Ngoài ra, CRISPR/Cas còn có thể được sử dụng trong việc tạo ra các loại động vật và thực vật biến đổi gen với hiệu quả cao. Nhiều kết quả khả quan ban đầu của việc sử dụng hệ thống này cũng đã được công bố trên các tạp chí uy tín trên thế giới.
Tin bài: Lã Văn Hiền
Người đăng: Bùi Đình Lãm
Tài liệu tham khảo:
6. Harrison M.M et al., 2014. A CRISPR view of development. Genes Dev 28, 1,859–1,872.
7. Gilbert L.A et al., 2013. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell 154, 442–451.
8. Chen B et al., 2013. Dynamic imaging of genomic loci in living human cells by an optimized CRISPR/Cas system. Cell 155, 1,479–1,491.
9. Qi L.S et al., 2013. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell, 152, 1173–1183.
10. Ran F.A et al., 2013. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell, 154, 1380–1389.
11. Gasiunas G et al., 2012. Cas9–crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc. Natl. Acad. Sci. USA, 109, E2579–2586.
12. Doudna J.A and Charpentier E., 2014. Genome editing. The new frontier of genome engineering with CRISPR-Cas9. Science 346(6213), 1258096. DOI: 10.1126/science.1258096.

| Đang online | 637 |
| Hôm nay | 2266 |
| Hôm qua | 3860 |
| Tuần này | 29543 |
| Tuần trước | 38389 |
| Tháng này | 3446050 |
| Tháng trước | 5230891 |
| Tất cả | 51666011 |
Lượt truy cập: 51666021
Đang online: 647
Ngày hôm qua: 3860
Phụ trách kỹ thuật: 0987. 008. 333